Inmunología Humana
-
INTRODUCCIÓN AL SISTEMA INMUNE HUMANOIntroducción. Conceptos básicos10 Temas
-
Introducción Inmunología
-
Funciones sistema inmune y barreras de defensa
-
Inmunidad innata vs adaptativa
-
Respuesta humoral vs celular
-
Respuesta adaptativa primaria vs secundaria
-
Características de la respuesta inmunitaria adaptativa
-
Concepto de antígeno
-
La respuesta inmunitaria específica es clonal
-
Revisión de los componentes del sistema inmunitario
-
Patología general del sistema inmunitario
-
Introducción Inmunología
-
Células del sistema inmune y diferenciación celular6 Temas
-
Hematopoyesis
-
Las células sanguíneas (I): Granulocitos y Mastocitos
-
Las células sanguíneas (II): Plaquetas, Eritrocitos, Monocitos y Macrófagos
-
Las células sanguíneas (III): Linfocitos (T y B), Células plasmáticas y LGL (Linfocitos NK)
-
Las células sanguíneas (IV): Células dendríticas y proporciones relativas de los tipos celulares
-
Funciones y gestión de receptores para antígenos
-
Hematopoyesis
-
Tejidos del sistema inmune: órganos linfoides 1º y 2º3 Temas
-
Células y mecanismos de la inmunidad innata (I): macrófagos, receptores y mecanismos efectores5 Temas
-
Células y mecanismos de la inmunidad innata (II): linfocitos NK, receptores y mecanismos efectores4 Temas
-
MOLÉCULAS IMPLICADAS EN EL RECONOCIMIENTO DE ANTÍGENOEl receptor de antígeno del linfocito B6 Temas
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
Inmunoglobulinas (II): Clases, formas, variabilidad y tejidos
-
Inmunoglobulinas (III): Enlaces antígeno-anticuerpo y funciones
-
Inmunoglobulinas (IV): Receptores para inmunoglobulinas
-
Inmunoglobulinas (V): Procesamiento de RNA, receptor del linfocito B, complejo co-receptor y activación célula B
-
Inmunoglobulinas (VI): Desarrollo / Diferenciación células B
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
El receptor de antígeno del linfocito T4 Temas
-
Mecanismos de generación de la diversidad de linfocitos T y B9 Temas
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
Reordenamiento de los genes
-
Mecanismos de amplificación de la diversidad
-
Expresión de las inmunoglobulinas en el BCR
-
Maduración de los linfocitos B
-
La cooperación T-B
-
Anomalías/Problemas en el proceso (CLÍNICA)
-
Maduración de los linfocitos B
-
Variabilidad en el repertorio del TcR en linfocitos T
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
El complejo principal de histocompatibilidad (I): estructura proteica, genética y nomenclatura3 Temas
-
El complejo principal de histocompatibilidad (II): Procesamiento y presentación de antígeno, polimorfismo y aplicaciones clínicas5 Temas
-
MOLÉCULAS ACCESORIAS DE LA RESPUESTA INMUNEEl sistema del complemento y sus receptores (I): vía clásica y vía alternativa4 Temas
-
El sistema del complemento y sus receptores (II): vía de las lectinas, vía lítica y regulación3 Temas
-
Moléculas implicadas en la comunicación intercelular (I): citocinas y sus receptores5 Temas
-
Moléculas implicadas en la comunicación intercelular (II): moléculas de adhesión y sus ligandos3 Temas
-
EL SISTEMA INMUNE EN ACCIÓN BLOQUEGeneración de linfocitos T efectores4 Temas
-
Generación de linfocitos B efectores7 Temas
-
Sistema Inmune asociado a mucosas (MALT)9 Temas
-
Introducción MALT
-
Estructura del tejido linfoide asociado a la mucosa intestinal
-
Recirculación y migración selectiva de linfocitos al intestino
-
Barrera epitelial intestinal: Función Inmunológica
-
Receptores para el reconocimiento de patrones moleculares
-
Inmunoglobulinas Poliméricas: IgM + IgA
-
Vías de entrada del antígeno en el intestino
-
Diferenciación de los linfocitos T efectores MALT
-
Tolerancia Oral ante antígenos solubles
-
Introducción MALT
-
La respuesta inmune (I): inmunidad innata e inflamación aguda8 Temas
-
Barreras no específicas frente a infección por microorganismos patógenos
-
Principales leucocitos que paticipan en las inmunidad innata
-
La rección inflamatoria
-
Inflamación aguda
-
Sucesos del proceso inflamatorio
-
Citocinas producidas por macrófagos: efectos locales y sistémicos
-
Efectos de las citocinas en la inmunidad innata
-
Proteínas de fase aguda
-
Barreras no específicas frente a infección por microorganismos patógenos
-
La respuesta inmune (II): mecanismos de la inmunidad específica8 Temas
-
La respuesta inmune (III): respuesta frente a virus, bacterias y hongos, protozoos y helmintos9 Temas
-
Principales enfermedades infecciosas mortales
-
Patógenos infecciosos
-
Respuestas frente a bacterias extracelulares
-
Respuesta frente a bacterias intracelulares
-
Proceso de fagocitosis
-
Respuestas frente a protozoos y helmintos
-
Ciclo vital del plasmodium
-
Respuestas inmunitarias frente a virus
-
Mecanismos de evasión inmunitaria. Virales y bacterianos.
-
Principales enfermedades infecciosas mortales
-
REGULACIÓN e INTRODUCCIÓN A LA INMUNOPATOLOGÍARegulación de la respuesta inmune (I): regulación por moléculas8 Temas
-
Regulación de la respuesta inmune: Introducción
-
Regulación por moléculas: el complemento (I)
-
Proteínas reguladoras del complemento y proceso de regulación (II)
-
Proceso de regulación por complemento (III)
-
Regulación de los anticuerpos preformados
-
Regulación por anticuerpos: Redes idiotípicas
-
Regulación por moléculas de linfocitos T
-
Regulación a nivel bioquímico: ITAM frente a ITIM
-
Regulación de la respuesta inmune: Introducción
-
Regulación de la respuesta inmune (II): regulación por células y sistemas4 Temas
-
El sistema inmune a lo largo del ciclo vital: Inmunosenescencia6 Temas
-
Introducción a la inmunopatología13 Temas
-
Conceptos básicos
-
Inmunodeficiencias
-
Las Inmunodeficiencias (II): Déficits de Complemento
-
Las Inmunodeficiencias (III): Déficits de citocinas en receptores
-
Las Inmunodeficiencias (IV): Déficits de TLR's
-
Las Inmunodeficiencias (V): Déficits de función fagocitaria
-
Las Inmunodeficiencias (VI): Déficit de linfocitos B
-
Las Inmunodeficiencias (VII): Déficits de linfocitos T
-
Inmunodeficiencias primarias (IDP's): Clínica y Diagnóstico
-
Hipersensibilidades
-
Autoinmunidad
-
Trasplantes. Rechazo
-
Inmunidad frente a tumores
-
Conceptos básicos
-
Introducción a la Inmunoterapia8 Temas
-
Vacunas (I): Bases inmunológicas
-
Vacunas (II): Tipos de vacunas
-
Inmunoterapia frente a enfermedades autoinmunes
-
Anticuerpos monoclonales en la terapia del cáncer
-
Otras estrategias anti-tumorales
-
Tratamiento “clásico” de la patología alérgica
-
Inmunoterapia en el trasplante de órganos
-
Tratamiento de inmunodeficiencias primarias
-
Vacunas (I): Bases inmunológicas
Reordenamiento de los genes
Aquí tienes el temario explicado en el vídeo anterior. Si tienes alguna duda plantéala en el sistema de comentarios del final de la página.
Procesamiento de los genes de inmunoglobulinas
Los segmentos génicos que codifican las regiones variables de las inmunoglobulinas se encuentran muy separados en el genoma en todas las células del organismo, excepto en las del linaje B.
Según la disposición descrita de los genes en segmentos en distintos cromosomas, la producción de inmunoglobulinas es imposible si no ocurre nada más. La aproximación de estos genes se produce en la médula ósea durante el proceso de maduración de los linfocitos B y tiene lugar por un proceso de recombinación entre los diferentes segmentos génicos denominado recombinación somática. Este proceso, no solo participa en la generación de la diversidad, sino que es imprescindible para que se expresen las inmunoglobulinas, y por esta razón, solo los linfocitos B pueden producirlas.
Procesamiento de las cadenas pesadas
En las cadenas pesadas, se producen 2 recombinaciones somáticas. En un primer paso de re- combinación un gen D se combina nuevamente al azar con un gen J, quedando correlativos en el cromosoma 14. Después al azar también se combina un gen V con el fragmento D-J que ya se había formado previamente (Figuras 8.2 y 8.4).
En ambas recombinaciones, hay pérdidas de grandes secuencias de DNA, y como resultado, los 3 fragmentos V D y J que se hallaban separados al inicio, se hallan correlativos. Estos 3 fragmentos (V-D-J) forman un exón para el dominio variable de la cadena pesada. El resto de dominios constantes será codificado por un gen C (hay 9 genes C, uno para cada clase y subclase de inmunoglobulina).
Procesamiento de las cadenas ligeras
En el caso de la cadena ligera, el mecanismo de recombinación somática se produce una vez (entre un fragmento V y uno J) (Figuras 8.3 y 8.4). En cuanto a la cadena κ, un gen V y un gen J (separados en el genoma) se combinan al azar, generando un exón que cuando se traduzca a proteína codificará para el dominio variable de su cadena ligera. Para el dominio constante no hay problema pues sólo existe un gen C. En el caso de la cadena λ ocurre lo mismo pero se ex- presa el gen C que sigue al J elegido al azar. De esta forma se elimina un gran segmento de DNA circular.
Este fenómeno de reordenamiento de genes ocurre durante la maduración del linfocito B en la médula ósea. Esta recombinación posibilita la síntesis de un RNA mensajero con los segmentos V, J y C necesarios, que se alinearán por escisión intrónica (se eliminan grandes segmentos de RNA en el procesamiento).
Mecanismo de recombinación somática
Para que sea posible el mecanismo de recombinación somática, existen dos elementos imprescindibles: las recombinasas y las secuencias señalizadoras de la recombinación (RSS).
Las recombinasas (Figura 8.4) son enzimas específicas que forman heterodímeros, y se cono- cen como RAG-1 y RAG-2. Por ejemplo se unen en el caso de λ a un gen V (V3) y a un gen J (J2).
Las secuencias señalizadoras de la recombinación (RSS) (Figura 8.4) son secuencias de nucleótidos muy conservadas, que flanquean los segmentos génicos. Estas señales están formadas por una secuencia de 7 pares de bases (heptámero) y otra de 9 (nonámero) separadas por DNA espaciador no conservada de 12 o 23 pb.
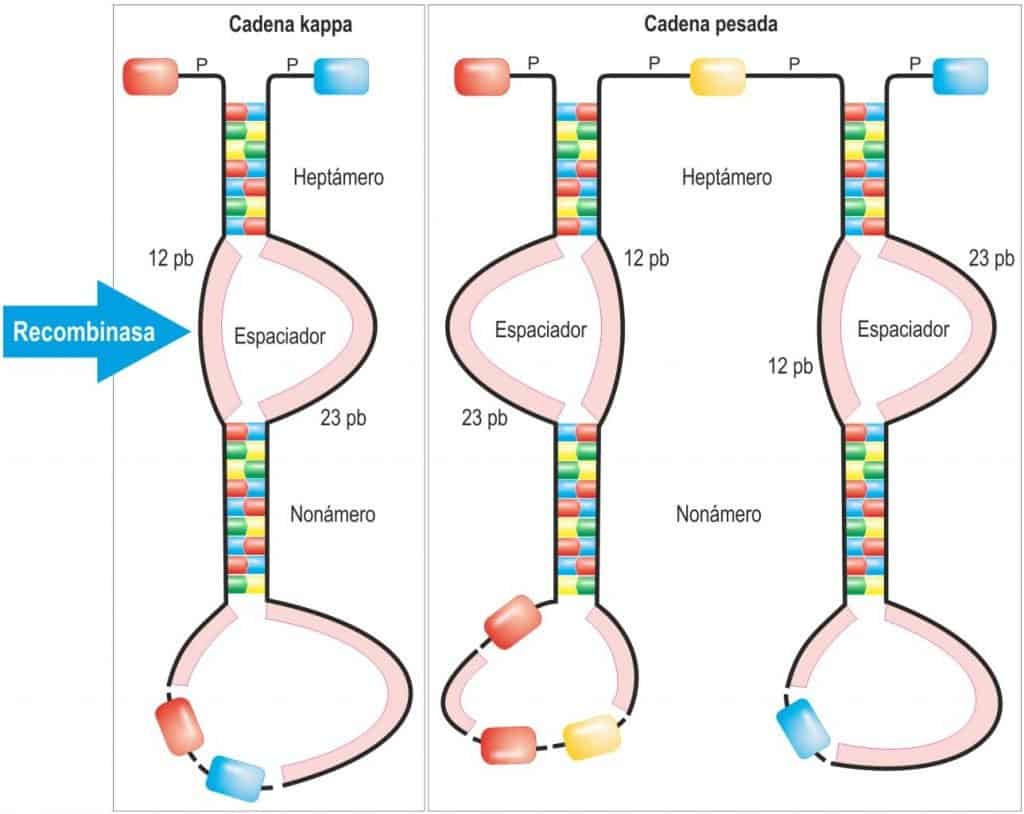
(Reproducido de Regueiro J.R., López C., González S. & Martínez E. (2011) Inmunología. Biología y Patología del Sistema Inmune. (4ª Ed.) Editorial Médica Panamericana, Madrid.)
Existen RSS posteriormente a cada fragmento V y previamente a cada segmento J. Los fragmentos D tienen RSS previos y posteriores, ya que participarán en 2 recombinaciones somáticas (la primera por detrás al combinarse con un segmento J, y la segunda por adelante cuando lo hace con un segmento V.
Las secuencias de los heptámeros y nonámeros de las RSS son complementarias unas a otras de modo que las secuencias posteriores del segmento V serán complementarias a las previas de segmento D. Y las posteriores del segmento D serán complementarias a las previas del segmento J (este hecho permite la formación de palíndromos).
La longitud de las regiones espaciadoras parece jugar un papel importante para que el proceso de recombinación somática de realice de forma ordenada (VDJ), ya que ésta se produce única- mente entre un par heptámerononámero separado 12 pares de bases (pb) y otro par heptámerononámero separado 23 pb. Estas secuencias espaciadoras de 12 pb y 23 pb se encuentran situadas de forma alterna en los diferentes segmentos génicos.
Por ello en las cadenas pesadas, no puede producirse una recombinación VJ, ya que ambas regiones se encuentran flanqueadas por espaciadores de 23 pb. Pero sí pueden recombinarse con los segmentos D, ya que están flanqueados por espaciadores de 12 pb.
Las señales de recombinación son reconocidas por las enzimas especializadas (recombinasas) RAG–1 y RAG–2, que al aproximarse entre sí, son responsables de la aproximación al azar entre los fragmentos formando un bucle en el ADN, que se estabiliza por los palíndromos entre los heptámeros y nonámeros señalizadores. A continuación, las recombinasas, resuelven el bu- cle cortando, ligando los 2 fragmentos y escindiendo los flecos (en forma de ADN circular).Todo lo que estaba en el bucle se pierde y es irrecuperable. En este corte y empalme de los fragmentos, que tiene imprecisiones se arrastran nucleótidos al azar y se insertan a veces nu- cleótidos. Esto supone un mecanismo de aumento de la variedad de Igs, como veremos más adelante, pero también puede alterar el orden de lectura dando lugar a codones STOP prematuros o a proteínas afuncionales (abortivas).
Debido a que el sistema de reordenamiento tiene muchas imprecisiones, tanto en los linfocitos B como en los T pueden ocurrir errores al reordenar los fragmentos produciéndose proteínas abortivas (codón STOP o cambio en la pauta de lectura) al azar, sin utilidad alguna (reordenamientos no productivos) (Figura 8.5). Por ello, tanto en el procesamiento de cadenas pesa- das como ligeras pueden rescatarse reordenamientos iniciales no productivos (siempre y cuando queden fragmentos V D y J disponibles) y volver a reordenar los cromosomas correspondientes al azar hasta obtener reordenamientos productivos, por tanto el límite está en el número de fragmentos que queden. Una vez conseguido un reordenamiento productivo existe una señal que paraliza el proceso.
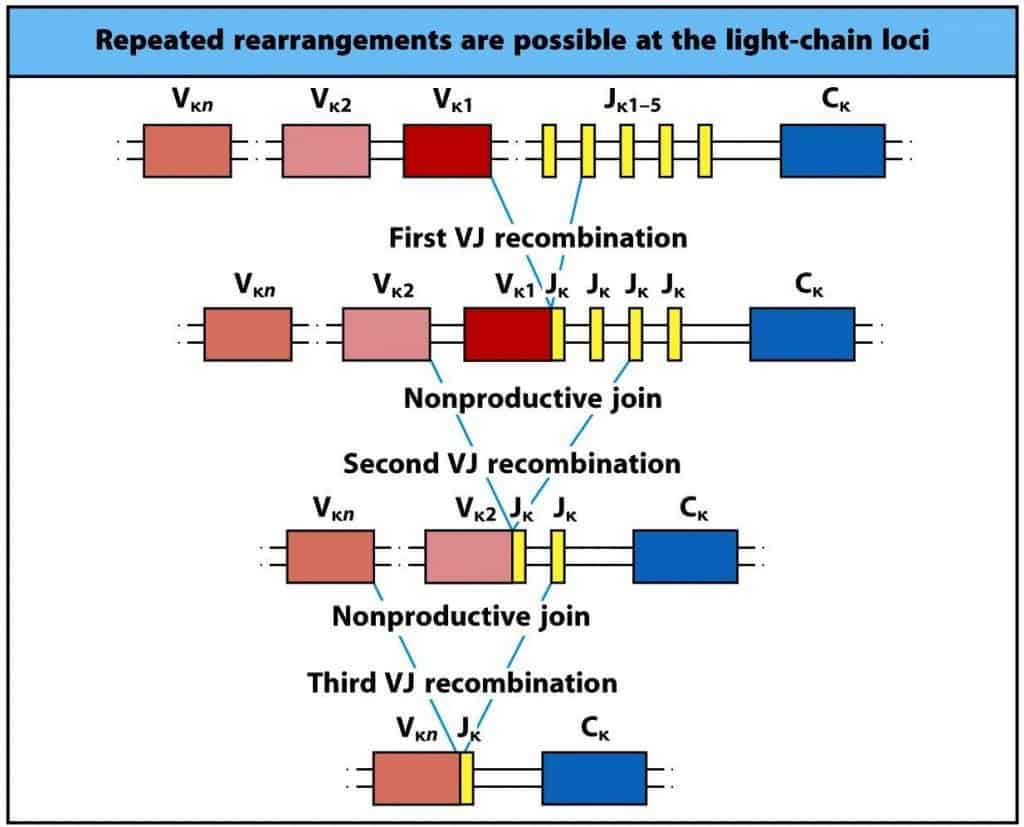
(Reproducido de Murphy K., Travers P. & Walport M. (2008) Janeway’s Immunobiology (7th Ed.) Garland Science, Nueva York.)

Responses