Inmunología Humana
-
INTRODUCCIÓN AL SISTEMA INMUNE HUMANOIntroducción. Conceptos básicos10 Temas
-
Introducción Inmunología
-
Funciones sistema inmune y barreras de defensa
-
Inmunidad innata vs adaptativa
-
Respuesta humoral vs celular
-
Respuesta adaptativa primaria vs secundaria
-
Características de la respuesta inmunitaria adaptativa
-
Concepto de antígeno
-
La respuesta inmunitaria específica es clonal
-
Revisión de los componentes del sistema inmunitario
-
Patología general del sistema inmunitario
-
Introducción Inmunología
-
Células del sistema inmune y diferenciación celular6 Temas
-
Hematopoyesis
-
Las células sanguíneas (I): Granulocitos y Mastocitos
-
Las células sanguíneas (II): Plaquetas, Eritrocitos, Monocitos y Macrófagos
-
Las células sanguíneas (III): Linfocitos (T y B), Células plasmáticas y LGL (Linfocitos NK)
-
Las células sanguíneas (IV): Células dendríticas y proporciones relativas de los tipos celulares
-
Funciones y gestión de receptores para antígenos
-
Hematopoyesis
-
Tejidos del sistema inmune: órganos linfoides 1º y 2º3 Temas
-
Células y mecanismos de la inmunidad innata (I): macrófagos, receptores y mecanismos efectores5 Temas
-
Células y mecanismos de la inmunidad innata (II): linfocitos NK, receptores y mecanismos efectores4 Temas
-
MOLÉCULAS IMPLICADAS EN EL RECONOCIMIENTO DE ANTÍGENOEl receptor de antígeno del linfocito B6 Temas
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
Inmunoglobulinas (II): Clases, formas, variabilidad y tejidos
-
Inmunoglobulinas (III): Enlaces antígeno-anticuerpo y funciones
-
Inmunoglobulinas (IV): Receptores para inmunoglobulinas
-
Inmunoglobulinas (V): Procesamiento de RNA, receptor del linfocito B, complejo co-receptor y activación célula B
-
Inmunoglobulinas (VI): Desarrollo / Diferenciación células B
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
El receptor de antígeno del linfocito T4 Temas
-
Mecanismos de generación de la diversidad de linfocitos T y B9 Temas
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
Reordenamiento de los genes
-
Mecanismos de amplificación de la diversidad
-
Expresión de las inmunoglobulinas en el BCR
-
Maduración de los linfocitos B
-
La cooperación T-B
-
Anomalías/Problemas en el proceso (CLÍNICA)
-
Maduración de los linfocitos B
-
Variabilidad en el repertorio del TcR en linfocitos T
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
El complejo principal de histocompatibilidad (I): estructura proteica, genética y nomenclatura3 Temas
-
El complejo principal de histocompatibilidad (II): Procesamiento y presentación de antígeno, polimorfismo y aplicaciones clínicas5 Temas
-
MOLÉCULAS ACCESORIAS DE LA RESPUESTA INMUNEEl sistema del complemento y sus receptores (I): vía clásica y vía alternativa4 Temas
-
El sistema del complemento y sus receptores (II): vía de las lectinas, vía lítica y regulación3 Temas
-
Moléculas implicadas en la comunicación intercelular (I): citocinas y sus receptores5 Temas
-
Moléculas implicadas en la comunicación intercelular (II): moléculas de adhesión y sus ligandos3 Temas
-
EL SISTEMA INMUNE EN ACCIÓN BLOQUEGeneración de linfocitos T efectores4 Temas
-
Generación de linfocitos B efectores7 Temas
-
Sistema Inmune asociado a mucosas (MALT)9 Temas
-
Introducción MALT
-
Estructura del tejido linfoide asociado a la mucosa intestinal
-
Recirculación y migración selectiva de linfocitos al intestino
-
Barrera epitelial intestinal: Función Inmunológica
-
Receptores para el reconocimiento de patrones moleculares
-
Inmunoglobulinas Poliméricas: IgM + IgA
-
Vías de entrada del antígeno en el intestino
-
Diferenciación de los linfocitos T efectores MALT
-
Tolerancia Oral ante antígenos solubles
-
Introducción MALT
-
La respuesta inmune (I): inmunidad innata e inflamación aguda8 Temas
-
Barreras no específicas frente a infección por microorganismos patógenos
-
Principales leucocitos que paticipan en las inmunidad innata
-
La rección inflamatoria
-
Inflamación aguda
-
Sucesos del proceso inflamatorio
-
Citocinas producidas por macrófagos: efectos locales y sistémicos
-
Efectos de las citocinas en la inmunidad innata
-
Proteínas de fase aguda
-
Barreras no específicas frente a infección por microorganismos patógenos
-
La respuesta inmune (II): mecanismos de la inmunidad específica8 Temas
-
La respuesta inmune (III): respuesta frente a virus, bacterias y hongos, protozoos y helmintos9 Temas
-
Principales enfermedades infecciosas mortales
-
Patógenos infecciosos
-
Respuestas frente a bacterias extracelulares
-
Respuesta frente a bacterias intracelulares
-
Proceso de fagocitosis
-
Respuestas frente a protozoos y helmintos
-
Ciclo vital del plasmodium
-
Respuestas inmunitarias frente a virus
-
Mecanismos de evasión inmunitaria. Virales y bacterianos.
-
Principales enfermedades infecciosas mortales
-
REGULACIÓN e INTRODUCCIÓN A LA INMUNOPATOLOGÍARegulación de la respuesta inmune (I): regulación por moléculas8 Temas
-
Regulación de la respuesta inmune: Introducción
-
Regulación por moléculas: el complemento (I)
-
Proteínas reguladoras del complemento y proceso de regulación (II)
-
Proceso de regulación por complemento (III)
-
Regulación de los anticuerpos preformados
-
Regulación por anticuerpos: Redes idiotípicas
-
Regulación por moléculas de linfocitos T
-
Regulación a nivel bioquímico: ITAM frente a ITIM
-
Regulación de la respuesta inmune: Introducción
-
Regulación de la respuesta inmune (II): regulación por células y sistemas4 Temas
-
El sistema inmune a lo largo del ciclo vital: Inmunosenescencia6 Temas
-
Introducción a la inmunopatología13 Temas
-
Conceptos básicos
-
Inmunodeficiencias
-
Las Inmunodeficiencias (II): Déficits de Complemento
-
Las Inmunodeficiencias (III): Déficits de citocinas en receptores
-
Las Inmunodeficiencias (IV): Déficits de TLR's
-
Las Inmunodeficiencias (V): Déficits de función fagocitaria
-
Las Inmunodeficiencias (VI): Déficit de linfocitos B
-
Las Inmunodeficiencias (VII): Déficits de linfocitos T
-
Inmunodeficiencias primarias (IDP's): Clínica y Diagnóstico
-
Hipersensibilidades
-
Autoinmunidad
-
Trasplantes. Rechazo
-
Inmunidad frente a tumores
-
Conceptos básicos
-
Introducción a la Inmunoterapia8 Temas
-
Vacunas (I): Bases inmunológicas
-
Vacunas (II): Tipos de vacunas
-
Inmunoterapia frente a enfermedades autoinmunes
-
Anticuerpos monoclonales en la terapia del cáncer
-
Otras estrategias anti-tumorales
-
Tratamiento “clásico” de la patología alérgica
-
Inmunoterapia en el trasplante de órganos
-
Tratamiento de inmunodeficiencias primarias
-
Vacunas (I): Bases inmunológicas
Mecanismos de amplificación de la diversidad
Aquí tienes el temario explicado en el vídeo anterior. Si tienes alguna duda plantéala en el sistema de comentarios del final de la página.
Mecanismos de amplificación del repertorio
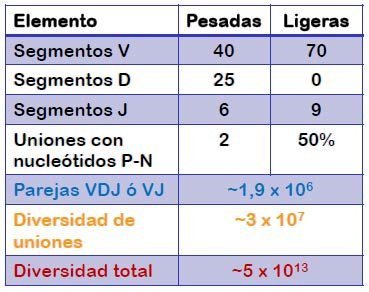
La diversidad de las inmunoglobulinas, deriva de la variabilidad de las cadenas ligeras y pesa- das, por una parte, y por otra de las diversas posibilidades de unirse entre sí estas cadenas. De esta forma obtendríamos una diversidad potencial de inmunoglobulinas de 4,5 millones. Sin embargo, se sabe que nuestro repertorio linfocitario alcanza el orden de 1011 (Figura 8.10), por ello existen dos mecanismos adicionales: diversidad de uniones, e hipermutaciones somáticas.
Diversidad de uniones
Estos mecanismos tienen lugar en el momento del reordenamiento genético durante la maduración del linfocito B en la médula ósea. Se producen porque la recombinación somática VDJ (en cadenas pesadas) o VJ (en cadenas ligeras) no es un mecanismo de alta precisión. De modo que el corte y empalme entre los diferentes fragmentos no es siempre igual, aunque participen los mismos fragmentos. Constituye un mecanismo de diversidad debido a varias causas:
- La propia flexibilidad en las uniones (Figura 8.6): El punto de corte del DNA en los 2 fragmentos puede variar, produciéndose codones alternativos. Es decir, la propia recombinasa no tiene por qué cortar los fragmentos siempre en el mismo punto. En función del punto de corte, y de entre qué nucleótidos se produzca la unión, puede cambiarse la pauta de lectura del DNA y con ello introducirse distintos aminoácidos en la proteína; aunque no todas las uniones dan lugar a reordenamientos VJ o VDJ productivos, puesto que pueden introducirse codones de terminación tempranos (Figura 8.7).
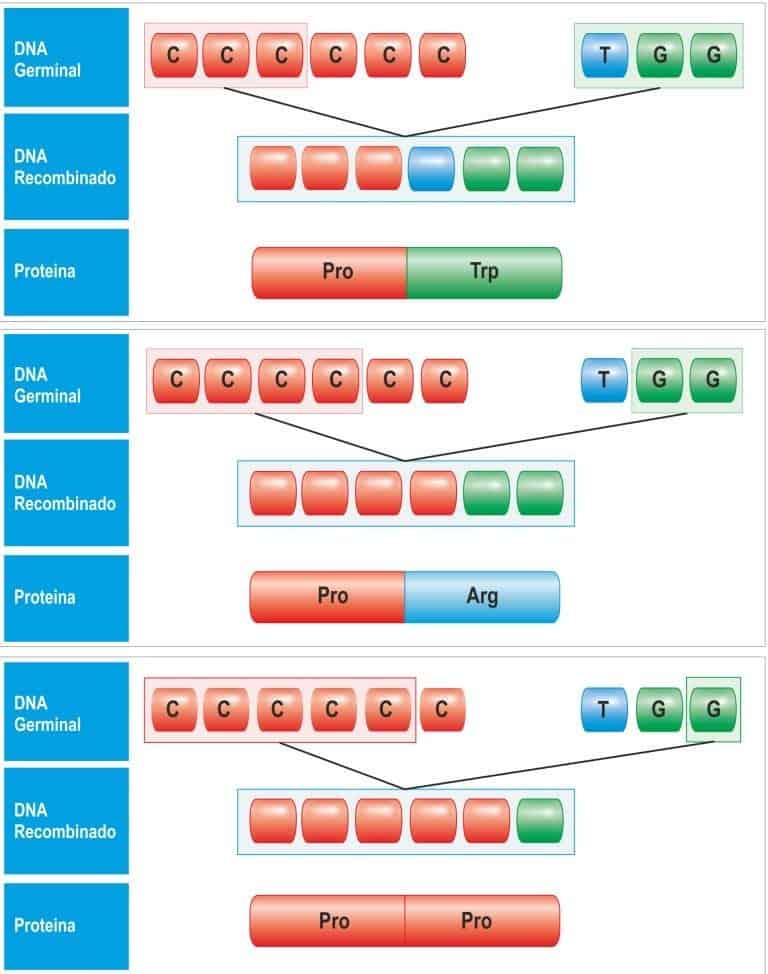
(Reproducido de Regueiro J.R., López C., González S. & Martínez E. (2011) Inmunología. Biolo- gía y Patología del Sistema Inmune. (4ª Ed.) Editorial Médica Panamericana, Madrid.)
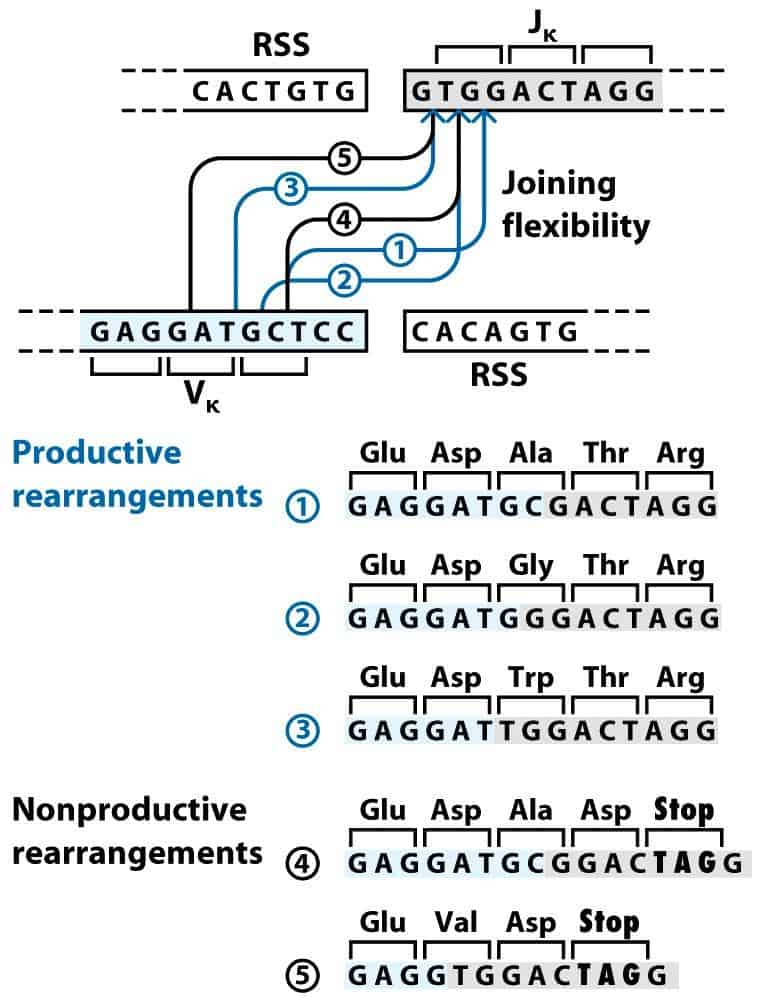
(Reproducido de Kindt T.J., Goldsby R.A., Osborne B.A. & Palacios R. (2007) Inmunología de Kuby (6ª Ed.) McGraw-Hill Interamericana, Madrid.)
- La adición de nucleótidos extra, que se efectúa en 2 procesos distintos:
- Los nucleótidos arrastrados en los reordenamientos, tras la escisión del DNA monocatenario, y que quedan colgando en el extremo, se vuelven para formar una estructura en horquilla (Figura 8.8-A) que protege al DNA. Cuando se rompe dicha horquilla, estos nucleótidos quedan “colgando” y la acción de enzimas reparadoras añade los nucleótidos complementarios a este filamento, generándose una secuencia palindrómica por lo que a estos nucleótidos se les llama nucleótidos P. En este proceso los nucleótidos P son arrastrados al azar tan- to en las cadenas ligeras como en las pesadas (y son responsables de un gran incremento en la variabilidad, pero también de posibles cambios en la pauta de lectura que darían lugar a proteínas abortivas).
- Pero además, el enzima deoxinucleotidil transferasa terminal (TdT) puede añadir unos pocos nucleótidos (en número variable) al azar sin molde en las zonas de unión. (Figura 8.8-B). Su incorporación genera una considerable diversidad. A estos nucleótidos se les denomina nucleótidos N. Esta adición de nucleótidos al azar por la TdT sucede siempre y en 2 sitios (entre los fragmentos D y J y entre los fragmentos V y D) en las cadenas pesadas. Sin embargo, en la cadena ligera sucede en un solo sitio (entre el fragmento V y el J) y solamente en el 50% de las ocasiones. Se ha observado que la longitud de nucleótidos N puede alcanzar hasta un número de 15.
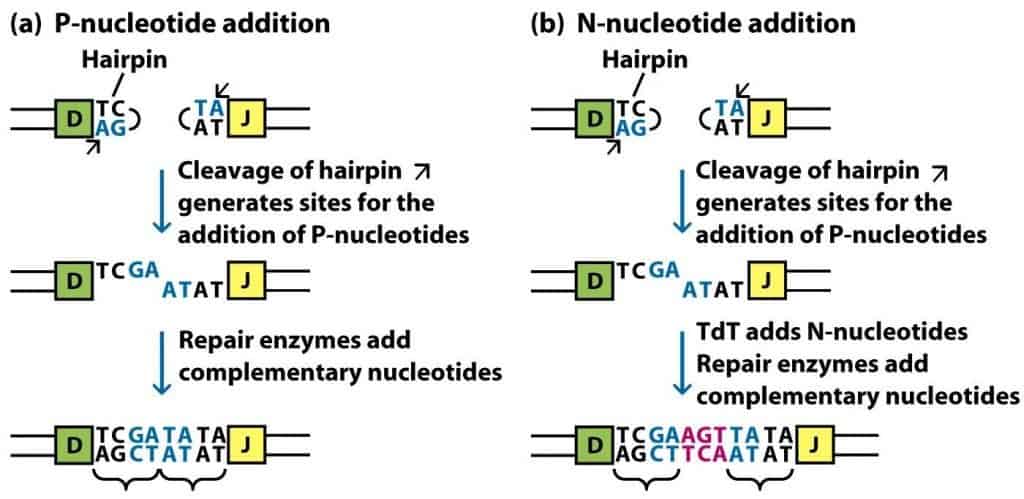
(Reproducido de Kindt T.J., Goldsby R.A., Osborne B.A. & Palacios R. (2007) Inmunología de Kuby (6ª Ed.) McGraw-Hill Interamericana, Madrid.)
Hipermutación somática
Este mecanismo inductor de variabilidad actúa en los órganos linfoides secundarios, una vez que un linfocito B maduro se ha activado en respuesta a un antígeno. Experimentalmente se ha podido comprobar que al introducir un antígeno en un ratón al cabo de unos días aparecen mutaciones en las regiones variables tanto de las cadenas pesadas como de las ligeras (Figura 8.9). Después, si existe inmunización secundaria se observan aún muchas más mutaciones en las regiones variables. Las mutaciones puntuales introducidas en este proceso pueden o no alterar la secuencia de aminoácidos de la región variable.
Los cambios que introducen modificaciones en la secuencia proteica (mutaciones productivas) se concentran en las regiones codificantes de las regiones hipervariables (CDRs, de interacción con el antígeno) mientras que las que no modifican la secuencia (mutaciones silenciosas) suelen producirse a nivel de las regiones flanqueantes. Este proceso se produce al azar en el momento en que la célula B responde al antígeno.
El resultado puede ser nefasto, dando lugar a codones de terminación que generen proteínas truncadas, o codificándose inmunoglobulinas que no reconocen al antígeno. En estos casos, los linfocitos B no progresan.
Sin embargo también existe la posibilidad de que se obtengan como consecuencia de la mutación anticuerpos más afines, que permitan un mejor reconocimiento del antígeno. Es por esto que este mecanismo también se denomina maduración de afinidad. Estos clones serán seleccionados, lo que permitirá un refinamiento de la respuesta inmune.
Este sistema de amplificación de repertorio no se incluye
en el cálculo de receptores distintos que se pueden generar, sin embargo es un
mecanismo más para la generación de diversidad.
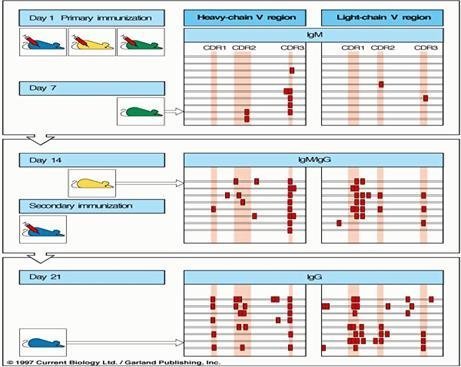
(Reproducido de Murphy K., Travers P. & Walport M. (2008) Janeway’s Immunobiology (7th Ed.) Garland Science, Nueva York.)

Responses