Inmunología Humana
-
INTRODUCCIÓN AL SISTEMA INMUNE HUMANOIntroducción. Conceptos básicos10 Temas
-
Introducción Inmunología
-
Funciones sistema inmune y barreras de defensa
-
Inmunidad innata vs adaptativa
-
Respuesta humoral vs celular
-
Respuesta adaptativa primaria vs secundaria
-
Características de la respuesta inmunitaria adaptativa
-
Concepto de antígeno
-
La respuesta inmunitaria específica es clonal
-
Revisión de los componentes del sistema inmunitario
-
Patología general del sistema inmunitario
-
Introducción Inmunología
-
Células del sistema inmune y diferenciación celular6 Temas
-
Hematopoyesis
-
Las células sanguíneas (I): Granulocitos y Mastocitos
-
Las células sanguíneas (II): Plaquetas, Eritrocitos, Monocitos y Macrófagos
-
Las células sanguíneas (III): Linfocitos (T y B), Células plasmáticas y LGL (Linfocitos NK)
-
Las células sanguíneas (IV): Células dendríticas y proporciones relativas de los tipos celulares
-
Funciones y gestión de receptores para antígenos
-
Hematopoyesis
-
Tejidos del sistema inmune: órganos linfoides 1º y 2º3 Temas
-
Células y mecanismos de la inmunidad innata (I): macrófagos, receptores y mecanismos efectores5 Temas
-
Células y mecanismos de la inmunidad innata (II): linfocitos NK, receptores y mecanismos efectores4 Temas
-
MOLÉCULAS IMPLICADAS EN EL RECONOCIMIENTO DE ANTÍGENOEl receptor de antígeno del linfocito B6 Temas
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
Inmunoglobulinas (II): Clases, formas, variabilidad y tejidos
-
Inmunoglobulinas (III): Enlaces antígeno-anticuerpo y funciones
-
Inmunoglobulinas (IV): Receptores para inmunoglobulinas
-
Inmunoglobulinas (V): Procesamiento de RNA, receptor del linfocito B, complejo co-receptor y activación célula B
-
Inmunoglobulinas (VI): Desarrollo / Diferenciación células B
-
Inmunoglobulinas (I): Formas en la naturaleza, estructura y dominio básico
-
El receptor de antígeno del linfocito T4 Temas
-
Mecanismos de generación de la diversidad de linfocitos T y B9 Temas
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
Reordenamiento de los genes
-
Mecanismos de amplificación de la diversidad
-
Expresión de las inmunoglobulinas en el BCR
-
Maduración de los linfocitos B
-
La cooperación T-B
-
Anomalías/Problemas en el proceso (CLÍNICA)
-
Maduración de los linfocitos B
-
Variabilidad en el repertorio del TcR en linfocitos T
-
Genes para las cadenas pesadas y ligeras: Diversidad potencial
-
El complejo principal de histocompatibilidad (I): estructura proteica, genética y nomenclatura3 Temas
-
El complejo principal de histocompatibilidad (II): Procesamiento y presentación de antígeno, polimorfismo y aplicaciones clínicas5 Temas
-
MOLÉCULAS ACCESORIAS DE LA RESPUESTA INMUNEEl sistema del complemento y sus receptores (I): vía clásica y vía alternativa4 Temas
-
El sistema del complemento y sus receptores (II): vía de las lectinas, vía lítica y regulación3 Temas
-
Moléculas implicadas en la comunicación intercelular (I): citocinas y sus receptores5 Temas
-
Moléculas implicadas en la comunicación intercelular (II): moléculas de adhesión y sus ligandos3 Temas
-
EL SISTEMA INMUNE EN ACCIÓN BLOQUEGeneración de linfocitos T efectores4 Temas
-
Generación de linfocitos B efectores7 Temas
-
Sistema Inmune asociado a mucosas (MALT)9 Temas
-
Introducción MALT
-
Estructura del tejido linfoide asociado a la mucosa intestinal
-
Recirculación y migración selectiva de linfocitos al intestino
-
Barrera epitelial intestinal: Función Inmunológica
-
Receptores para el reconocimiento de patrones moleculares
-
Inmunoglobulinas Poliméricas: IgM + IgA
-
Vías de entrada del antígeno en el intestino
-
Diferenciación de los linfocitos T efectores MALT
-
Tolerancia Oral ante antígenos solubles
-
Introducción MALT
-
La respuesta inmune (I): inmunidad innata e inflamación aguda8 Temas
-
Barreras no específicas frente a infección por microorganismos patógenos
-
Principales leucocitos que paticipan en las inmunidad innata
-
La rección inflamatoria
-
Inflamación aguda
-
Sucesos del proceso inflamatorio
-
Citocinas producidas por macrófagos: efectos locales y sistémicos
-
Efectos de las citocinas en la inmunidad innata
-
Proteínas de fase aguda
-
Barreras no específicas frente a infección por microorganismos patógenos
-
La respuesta inmune (II): mecanismos de la inmunidad específica8 Temas
-
La respuesta inmune (III): respuesta frente a virus, bacterias y hongos, protozoos y helmintos9 Temas
-
Principales enfermedades infecciosas mortales
-
Patógenos infecciosos
-
Respuestas frente a bacterias extracelulares
-
Respuesta frente a bacterias intracelulares
-
Proceso de fagocitosis
-
Respuestas frente a protozoos y helmintos
-
Ciclo vital del plasmodium
-
Respuestas inmunitarias frente a virus
-
Mecanismos de evasión inmunitaria. Virales y bacterianos.
-
Principales enfermedades infecciosas mortales
-
REGULACIÓN e INTRODUCCIÓN A LA INMUNOPATOLOGÍARegulación de la respuesta inmune (I): regulación por moléculas8 Temas
-
Regulación de la respuesta inmune: Introducción
-
Regulación por moléculas: el complemento (I)
-
Proteínas reguladoras del complemento y proceso de regulación (II)
-
Proceso de regulación por complemento (III)
-
Regulación de los anticuerpos preformados
-
Regulación por anticuerpos: Redes idiotípicas
-
Regulación por moléculas de linfocitos T
-
Regulación a nivel bioquímico: ITAM frente a ITIM
-
Regulación de la respuesta inmune: Introducción
-
Regulación de la respuesta inmune (II): regulación por células y sistemas4 Temas
-
El sistema inmune a lo largo del ciclo vital: Inmunosenescencia6 Temas
-
Introducción a la inmunopatología13 Temas
-
Conceptos básicos
-
Inmunodeficiencias
-
Las Inmunodeficiencias (II): Déficits de Complemento
-
Las Inmunodeficiencias (III): Déficits de citocinas en receptores
-
Las Inmunodeficiencias (IV): Déficits de TLR's
-
Las Inmunodeficiencias (V): Déficits de función fagocitaria
-
Las Inmunodeficiencias (VI): Déficit de linfocitos B
-
Las Inmunodeficiencias (VII): Déficits de linfocitos T
-
Inmunodeficiencias primarias (IDP's): Clínica y Diagnóstico
-
Hipersensibilidades
-
Autoinmunidad
-
Trasplantes. Rechazo
-
Inmunidad frente a tumores
-
Conceptos básicos
-
Introducción a la Inmunoterapia8 Temas
-
Vacunas (I): Bases inmunológicas
-
Vacunas (II): Tipos de vacunas
-
Inmunoterapia frente a enfermedades autoinmunes
-
Anticuerpos monoclonales en la terapia del cáncer
-
Otras estrategias anti-tumorales
-
Tratamiento “clásico” de la patología alérgica
-
Inmunoterapia en el trasplante de órganos
-
Tratamiento de inmunodeficiencias primarias
-
Vacunas (I): Bases inmunológicas
Nomenclatura HLA y peculiaridades genéticas
Aquí tienes el temario explicado en el vídeo anterior. Si tienes alguna duda plantéala en el sistema de comentarios del final de la página.
Nomenclatura del sistema HLA
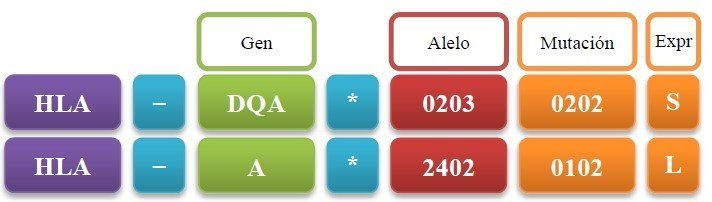
La nomenclatura de los antígenos del sistema HLA se realiza de forma estandarizada. El nombre comienza con las siglas HLA seguidas de un guión. A continuación, se indica el gen al que nos estamos refiriendo con la letra que lo identifica como de clase I (A, B, C, D, E, F, G) o de clase II (DP, DQ, DR). En caso de que sea de clase II, se debe especificar si es un gen que codifica la cadena alfa (A), o la cadena beta (B). En el caso de que haya varios genes para esa cadena, como en el caso de DRB, se debe especificar el número del gen. Una vez especificado el gen, se escribe un asterisco y, a continuación, el alelo de dicho gen presente en el cromosoma del individuo (Figura 9.8).
El alelo se localiza con precisión mediante un sistema de, al menos, cuatro números. La primera y segunda cifras indican el grupo alélico. La tercera y cuarta cifra indican el alelo concreto dentro de ese grupo. El sistema puede complementarse con dígitos que aportan más información: la quinta y sexta cifras se emplean para indicar mutaciones en exones, la séptima y octava cifras se emplean para indicar mutaciones en intrones. Finalmente, se puede añadir una letra mayúscula (L, N, Q, S) que especifica el nivel de expresión de la proteína.
Por ejemplo, el gen HLA-DRB3*0202 se refiere al gen que codifica la cadena beta de tipo tres para un antígeno HLA de clase II tipo DR; específicamente el segundo alelo del segundo grupo de este gen.
Mecanismos de variabilidad
Una serie de mecanismos de variabilidad hacen que sea extremadamente difícil encontrar dos individuos de la población cuyas células expresen la misma colección de antígenos HLA. Estos mecanismos son la poligenia, la codominancia, el polimorfismo y la complementación.
Todos estos mecanismos hacen de la combinación de antígenos de cada individuo una marca identificativa muy fiable, siendo las probabilidades de error son del orden de 1/106. Nuestras células presentan tantos y tan variados antígenos HLA en su superficie para que se puedan pre- sentar el mayor número posible de péptidos antigénicos diferentes y tener asegurado que tenga- mos linfocitos T con un TcR adecuado. Además hay que tener en cuenta que todos estos genes están muy próximos, en una región de 1cM del brazo corto del cromosoma 6. Esto hace que la tasa de recombinación sea muy baja y, consecuentemente, estos genes tienden a heredarse en bloque, formando el llamado haplotipo HLA. Esto, unido a la gran variabilidad ya explicada, hace muy útil el análisis de los antígenos HLA para diversas pruebas identificativas como las pruebas de paternidad.
Poligenia
La poligenia, o presencia de varios genes diferentes relacionados con las mismas funciones, asegura que cada individuo produzca una gran variedad de moléculas HLA. Por ejemplo, las moléculas HLA-A, B y C tienen la misma función y, sin embargo, todas ellas se sintetizan en cada una de nuestras células nucleadas (Figura 9.9).
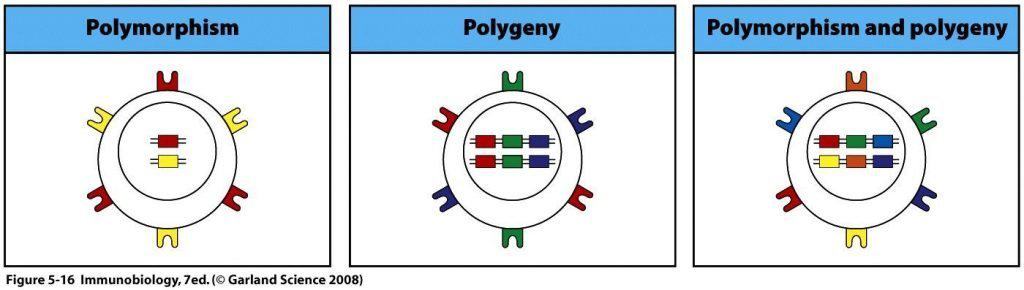
(Reproducido de Murphy K et al. (2008) Janeway’s Immunobiology (7th Ed.) Garland Science, Nueva York.)
Codominancia
Se expresan los genes de ambos cromosomas. Por ello en la membrana de las células se encuentran tanto los antígenos HLA heredados por vía materna, como los heredados por vía paterna (Figura 9.10). La poligenia y la codominancia tienen como consecuencia el hecho de que cual- quier célula nucleada del organismo puede presentar hasta seis antígenos HLA de clase I dife- rentes: A B y C heredados del padre; y A B y C heredados de la madre.
Polimorfismo
Los genes que codifican los antígenos HLA presentan un gran
número de alelos distintos. Es decir, hay muchas formas diferentes para cada
uno de los genes (muchas formas de A, muchas formas de B y muchas formas de C
para los HLA de clase I) y cada uno de nosotros expresamos dos de esas formas
en nuestras células, una de herencia materna y otra de herencia paterna.
Se pueden ordenar los genes del sistema HLA de acuerdo a su polimorficidad (Figura 9.11):
- HLA de clase I: HLA-B > HLA-A > HLA-C.
- HLA de clase II: HLA-DRB > HLA-DP > HLA-DQ. Dentro de los HLA-DRB el más polimórfico es HLA-DRB1.
Esta variabilidad no se reparte homogéneamente en todo el gen, sino que se concentra en unas regiones hipervariables, que son las que coinciden con los aminoácidos de la cavidad de unión de péptidos (Figura 9
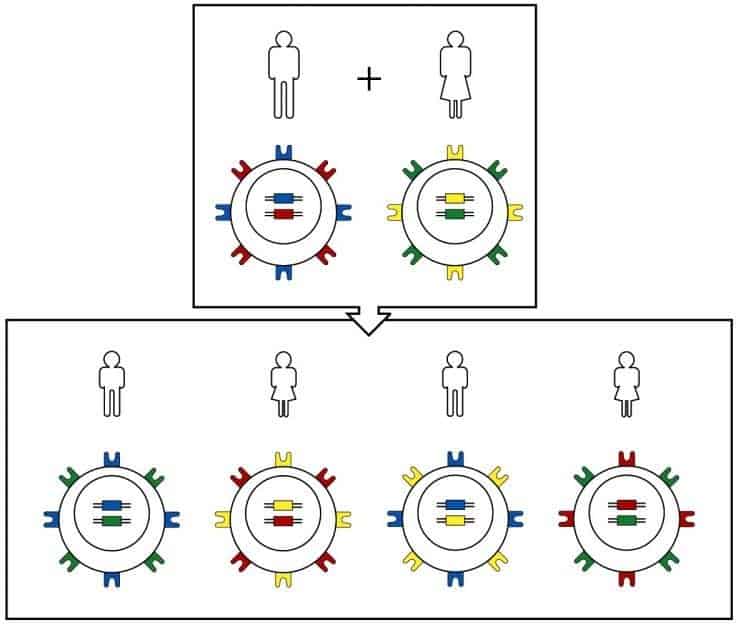
El MHC es tan polimórfico que es probable que casi todos los individuos sean heterocigotos para cada locus. En un individuo se expresan los alelos de ambos haplotipos MHC, y la expresión de estos se traduce en la presencia de sus productos en la superficie celular. (Reproducido de Murphy
K et al. (2008) Janeway’s Immunobiology (7th Ed.) Garland Science, Nueva York.)
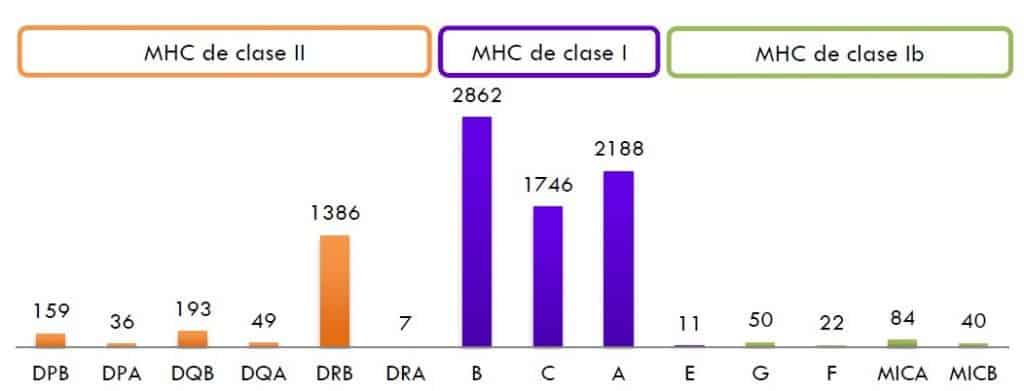
En el gráfico se muestran el número de alelos conocidos para cada locus HLA. Los loci más poli- mórficos son HLA-B y HLA-DRB. Los números que aparecen son meramente indicativos, ya que el descubrimiento de nuevos alelos es constante. La altura de las barras representa el número de alelos del sistema HLA asignados por el WHO Nomenclature Committee for Factors of the HLA System (actualizado en enero de 2013).
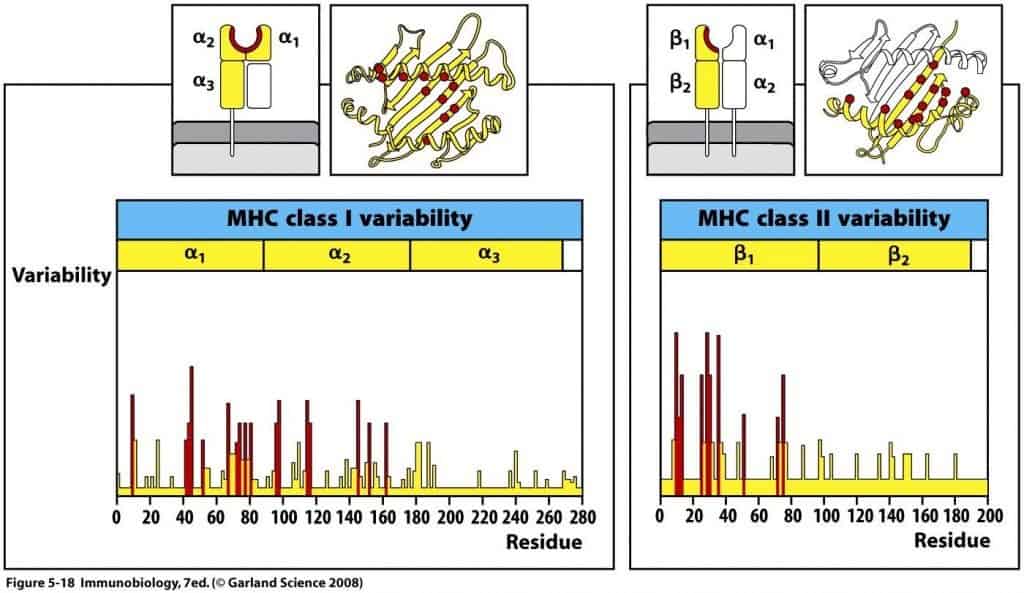
(Reproducido de Murphy K et al. (2008) Janeway’s Immunobiology (7th Ed.) Garland Science, Nueva York.)
Complementación
Existe un último mecanismo de variabilidad, presente tan sólo en los antígenos HLA de clase II, denominado complementación (Figura 9.13). Las moléculas HLA de clase II se componen de una cadena pesada beta y una cadena pesada alfa, que se sintetizan de forma independiente en el retículo plasmático. Esto hace que la unión de una cadena alfa con una cadena beta se produzca al azar, con dos posibles resultados:
- Complementación en cis: Una cadena alfa materna se une a una cadena beta materna o una cadena alfa paterna se une a una cadena beta materna.
- Complementación en trans: Una cadena alfa materna se une a una cadena beta paterna o una cadena alfa paterna se une a una cadena beta materna.
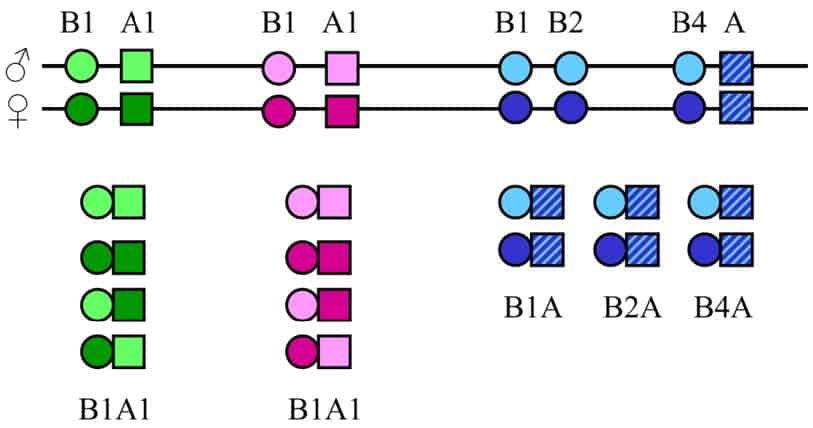
Fisiología del sistema HLA
La función fisiológica de las moléculas de HLA de clase I y HLA de clase II, consiste en presentar péptidos antígenicos, una vez que éste ha sido procesado, a los linfocitos Tc y Th respectivamente, dado que los TcR, al contrario que los BcR, no son capaces de reconocer el antígeno en su estado nativo.
Las moléculas de HLA también tienen una importante relevancia en otras condiciones no fisiológicas como: en el trasplante de órganos, para identificar individuos en medicina forense y también confieren susceptibilidad a padecer ciertas enfermedades autoinmunes. Las moléculas de HLA sólo se expresan en la superficie celular cuando se encuentran unidas al péptido, denominándose entonces complejo antigénico.
Cuando una molécula de HLA de clase I de la superficie de cualquier célula nucleada se une a su TcR apropiado, su cavidad de unión de péptidos contiene ya el péptido del antígeno. El correceptor del linfocito Tc, CD8, se asocia al dominio α3 de la molécula de HLA de clase I (Figura 9.14).
Cuando una molécula de HLA de clase II se expresa en la superficie de una célula presentadora de antígenos, con el péptido ya unido a su cavidad de unión de péptidos, y se une al TcR del linfocito Th, el correceptor, CD4, se unirá a su ligando natural que es el dominio β2 de la molécula HLA de clase II (Figura 9.14).
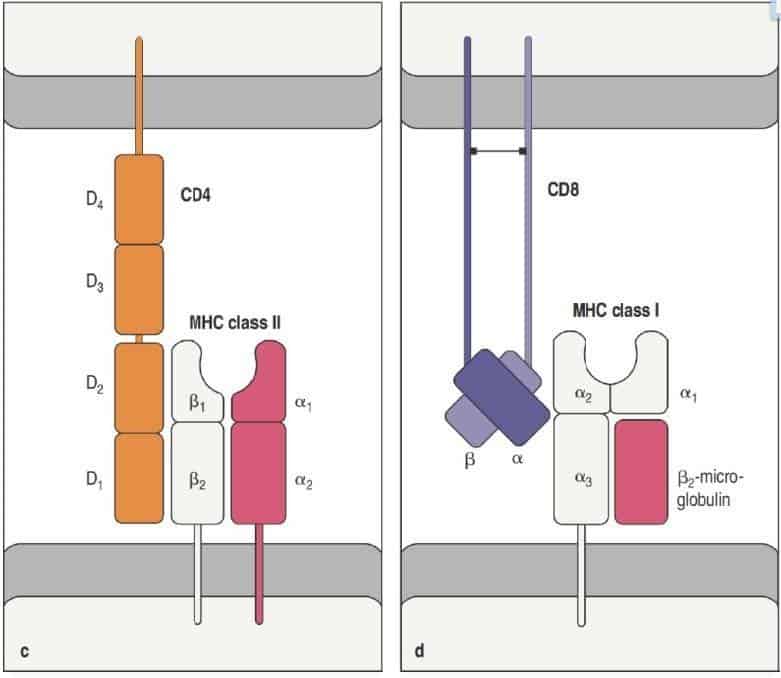
(Reproducido de Murphy K et al. (2012) Janeway’s Immunobiology (8th Ed.) Garland Science, Nueva York.)
Los correceptores CD4 y CD8 son glicoproteinas que facilitan la unión intercelular y además potenciarán la transmisión de señales al interior. Una vez que el complejo antigénico es reconocido por TcR, los linfocitos T comienzan a producir citocinas, que actúan como señales químicas. Las citocinas atraen más linfocitos T e inducen la proliferación de linfocitos B para que produzcan anticuerpos. Los anticuerpos se liberan a la circulación sanguínea para encontrar y unir más antígenos, de forma que los patógenos no se puedan reproducir y causar enfermedades.
(Reproducido de Regueiro J.R., López C., González S. & Martínez E. (2011) Inmunología. Biología y Patología del Sistema Inmune. (4ª Ed.) Editorial Médica Panamericana, Madrid.)
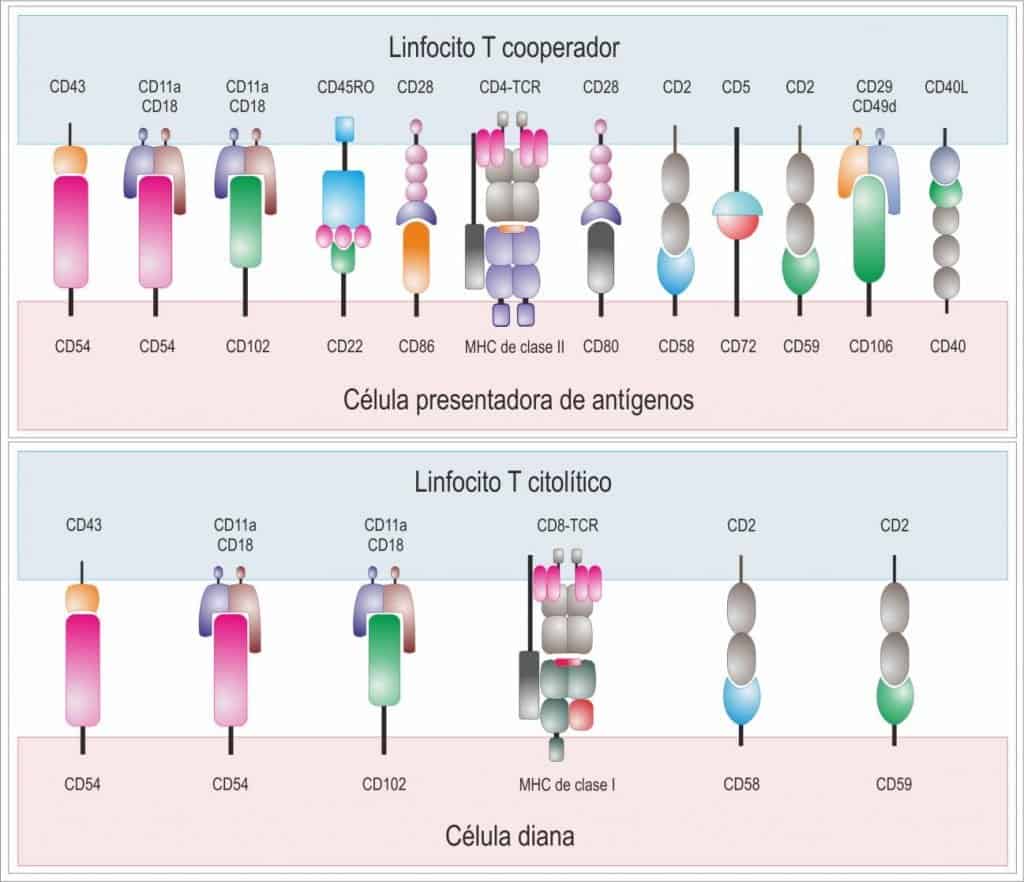
En muchos casos la unión del complejo antigénico con el TcR no es suficiente para dar lugar a una respuesta eficaz, requiriéndose la participación de una serie de moléculas accesorias, cuya función es la de contribuir al desarrollo de una respuesta inmune efectiva facilitando la interacción entre las distintas células. Se forma así una sinapsis inmunológica entre linfocitos T y la célula que presenta el antígeno, más intensa entre los linfocitos T cooperadores y las células presentadoras de antígenos que la que se produce entre los linfocitos T citotóxicos y sus células diana (Figura 9.15).
En la sinapsis inmunológica participan moléculas muy variadas, entre las que destacan:
- Receptores específicos de antígeno: BcR y TcR.
- Co-receptores CD4 y CD8.
- Moléculas presentadoras de antígeno: HLA de clase I y clase II.
- Receptores de moléculas adaptadoras (opsoninas): receptores para proteínas de complemento (CD21) y receptores para Fc de Inmunoglobulinas (CD16).
- Receptores para citocinas: para IL-1 (IL-1R ó CD121a) y para IL-2 (CD25).
- Moléculas de co-estimulación que intervienen en la activación y la diferenciación: CD28 (linfocitos T) y CD80 o CD86 (en la APC), CD19, CD20 (linfocitos B), CD2 (linfocitos T y NK), CD45 (marcador panleucocitario); CD40 (en la APC o linfocito B) y CD40L (en el linfocito T).
- Moléculas inducidas tras la activación: CD25, CD38 y CD69.
- Moléculas de adhesión celular: CD2, CD11(a,b,c), CD18.
- Marcadores de estado de la diferenciación celular: CD45R0 (memoria) o CD45RA (vírgenes).

Responses